oncotator是一款什么軟件
這篇文章主要介紹了oncotator是一款什么軟件,具有一定借鑒價(jià)值,感興趣的朋友可以參考下,希望大家閱讀完這篇文章之后大有收獲,下面讓小編帶著大家一起了解一下。
創(chuàng)新互聯(lián)主營(yíng)榆次網(wǎng)站建設(shè)的網(wǎng)絡(luò)公司,主營(yíng)網(wǎng)站建設(shè)方案,重慶APP軟件開發(fā),榆次h5微信小程序開發(fā)搭建,榆次網(wǎng)站營(yíng)銷推廣歡迎榆次等地區(qū)企業(yè)咨詢
目前,較為流行的突變注釋軟件有以下3種
ANNOVAR
SnpEff
Variant Effect Predictor(VEP)
這三款軟件適用范圍廣,可以注釋任何的基因組變異,無論是germline還是somatic variants。通用性強(qiáng)的同時(shí),帶來的問題就是針對(duì)腫瘤基因組研究而言,其注釋結(jié)果中缺乏腫瘤特異性的注釋內(nèi)容,而且其注釋結(jié)果為VCF格式,需要進(jìn)一步轉(zhuǎn)換為MAF格式才可以進(jìn)行腫瘤研究的下游分析,不夠便利。
為了解決這個(gè)問題,Broad Institute的科學(xué)家開發(fā)了一款名為oncotator的注釋軟件,專門針對(duì)腫瘤基因組研究進(jìn)行設(shè)計(jì),其注釋結(jié)果可以直接輸出成MAF格式,官網(wǎng)如下
https://software.broadinstitute.org/cancer/cga/oncotator
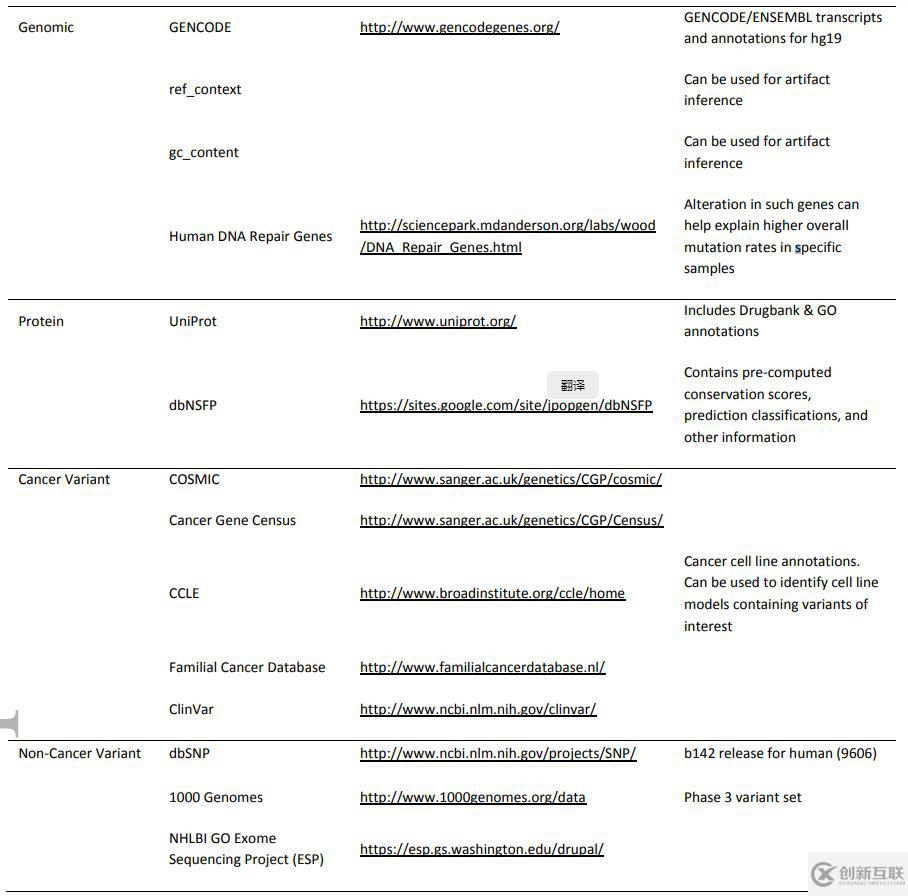
該軟件集成了14種不同來源的注釋信息,分成以下4大類別
Genomic Annotations
Protein Annotations
Cancer Variant Annotations
Non-Cancer Variant Annotations
不同類別對(duì)應(yīng)的數(shù)據(jù)庫展示如下
該軟件的源代碼保存在github上,網(wǎng)址如下
https://github.com/broadinstitute/oncotator/releases
同時(shí)該軟件依賴一個(gè)整合好的數(shù)據(jù)庫,下載方式如下
wget -r "ftp://gsapubftp-anonymous@ftp.broadinstitute.org/bundle/oncotator/"
數(shù)據(jù)庫大小為17G, 軟件采用python進(jìn)行開發(fā),其安裝過程比較簡(jiǎn)單,基本用法如下
oncotator \
-v \
--db-dir /oncotator_v1_ds_Jan262015 \
--input_format=VCF \
--output_format=TCGAMAF \
input.vcf \
output.maf \
hg19db-dir參數(shù)指定下載的數(shù)據(jù)數(shù)據(jù)庫的路徑,最后三個(gè)參數(shù)對(duì)應(yīng)輸入文件,輸出文件和基因組版本,該軟件只支持hg19版本。
為了方便沒有編程經(jīng)驗(yàn)的科研工作者,同時(shí)也提供了web 服務(wù),網(wǎng)址如下
http://portals.broadinstitute.org/oncotator/
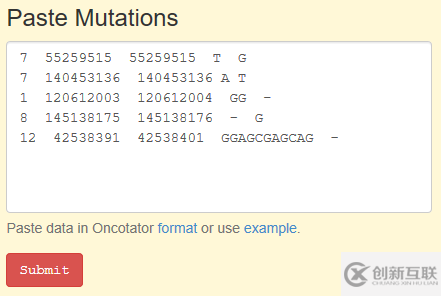
上傳tsv格式的突變數(shù)據(jù)即可,示意如下
結(jié)果展示如下
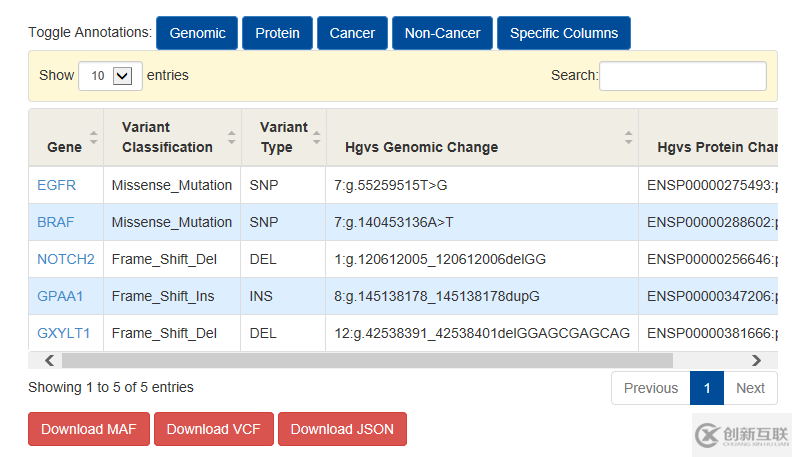
通過該軟件可以得到MAF格式的注釋文件,更加方便的用于下游的數(shù)據(jù)分析。
感謝你能夠認(rèn)真閱讀完這篇文章,希望小編分享的“oncotator是一款什么軟件”這篇文章對(duì)大家有幫助,同時(shí)也希望大家多多支持創(chuàng)新互聯(lián),關(guān)注創(chuàng)新互聯(lián)行業(yè)資訊頻道,更多相關(guān)知識(shí)等著你來學(xué)習(xí)!
網(wǎng)站欄目:oncotator是一款什么軟件
本文路徑:http://chinadenli.net/article14/jgjcge.html
成都網(wǎng)站建設(shè)公司_創(chuàng)新互聯(lián),為您提供網(wǎng)站營(yíng)銷、定制開發(fā)、網(wǎng)站內(nèi)鏈、商城網(wǎng)站、軟件開發(fā)、企業(yè)建站
聲明:本網(wǎng)站發(fā)布的內(nèi)容(圖片、視頻和文字)以用戶投稿、用戶轉(zhuǎn)載內(nèi)容為主,如果涉及侵權(quán)請(qǐng)盡快告知,我們將會(huì)在第一時(shí)間刪除。文章觀點(diǎn)不代表本網(wǎng)站立場(chǎng),如需處理請(qǐng)聯(lián)系客服。電話:028-86922220;郵箱:631063699@qq.com。內(nèi)容未經(jīng)允許不得轉(zhuǎn)載,或轉(zhuǎn)載時(shí)需注明來源: 創(chuàng)新互聯(lián)

- 網(wǎng)站策劃中8個(gè)小技巧讓你的網(wǎng)站脫穎而出 2021-01-12
- 如何利用網(wǎng)站策劃做好網(wǎng)站建設(shè)? 2014-12-20
- 教你如何做好網(wǎng)站策劃 2016-11-04
- 網(wǎng)站策劃方案絕不能忽視的幾個(gè)問題! 2021-04-28
- 網(wǎng)站策劃不能脫離實(shí)際 2016-11-02
- 網(wǎng)站策劃——網(wǎng)站建設(shè)的靈魂 2022-12-01
- 網(wǎng)站策劃?rùn)C(jī)構(gòu)議校園網(wǎng)站的意義 2022-12-08
- 網(wǎng)站建設(shè):電子商務(wù)網(wǎng)站策劃書如何制作 2021-02-24
- 濟(jì)寧網(wǎng)站建設(shè)-網(wǎng)站策劃需要做的具體工作 2021-11-27
- 網(wǎng)站策劃需要避免那些誤區(qū) 2016-11-02
- 創(chuàng)新互聯(lián)對(duì)網(wǎng)站策劃建設(shè)、運(yùn)營(yíng)維護(hù)、網(wǎng)站優(yōu)化含義介紹 2021-01-12
- 怎樣為客戶做好網(wǎng)站策劃 2016-11-04